Eine weitere Option für ein wenig Programmierung ist das Indigo Toolkit mit Python-, Java-, .NET- und C-Bibliotheken.
Das folgende Python-Beispiel verwendet das isomere SMILES für Albuterol, das ich von PubChem erhalten habe, und rendert 2D-Strukturen für drei Formen (eine mit nicht spezifizierter Stereochemie und die beiden Stereoisomere). Die einzelnen Strukturen sind mit ihren jeweiligen PubChem-CIDs gekennzeichnet.
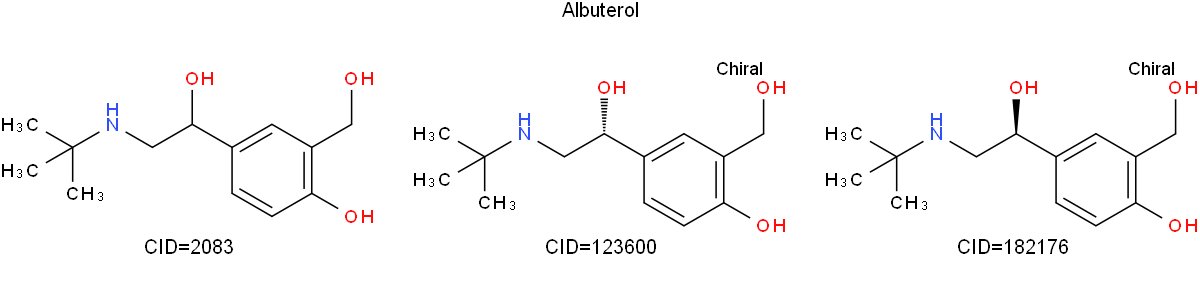
aus Indigo-Import * aus Indigo.Renderer-Import * Indigo = Indigo () Renderer = IndigoRenderer (Indigo) mols = {'2083': 'CC (C) (C) NCC (C1 = CC (= C (C = C1) O) CO) O', '123600': 'CC (C) (C) NC [C @@ H] (C 1 = CC (= C (C = C 1) O) CO) O ',' 182176 ':' CC (C) (C) NC [C @ H] (C 1 = CC (= C (C = C 1) ) O) CO) O '} array = indigo.createArray () für die Eingabe von mols.keys (): print (Schlüssel, mols [Schlüssel]) mol = indigo.loadMolecule (mols [Schlüssel]) s = "CID =" + key mol.setProperty ("grid-comment", s) array.arrayAdd (mol) indigo.setOption ("render-comment", "Albuterol") indigo.setOption ("render-comment-position", "top") indigo.setOption ("Render-Grid-Ränder", "40, 10") indigo.setOption ("Render-Grid-Titel-Offset", "5") indigo.setOption ("Render-Grid-Titel-Eigenschaft", "grid-comment") indigo.setOption ("Render-Hintergrundfarbe", 1.0, 1.0, 1.0) indigo.setOption ("Render-Atom-Farbeigenschaft", "Farbe") indigo.setOption ("Render-Färbung") ", True) indigo.setOption (" Renderbildgröße ", 1200, 300) renderer.renderGridToFile (Array, Keine, 3, "grid.png")